retro-enantio-Retrocyclin:
Aufgabe:
Stellen Sie Makakenretrozyklin (1HVZ) per PyMol dar
Mutieren Sie mithilfe des mutation-wizzard in PyMol die Aminosäuren hin zum
menschlichen retro-Retrozyklin. Die Position der Cysteine soll nicht geändert werden.
Bauen Sie daraus retro-enantio-Retrozyklin durch Umwandlung von D- in LAminosäuren.
Am Schluss: mit "sculpting" Bindungslängen und -winkel automatisch korrigieren
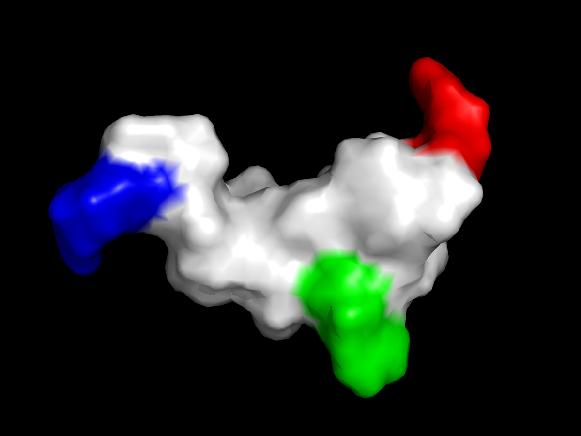
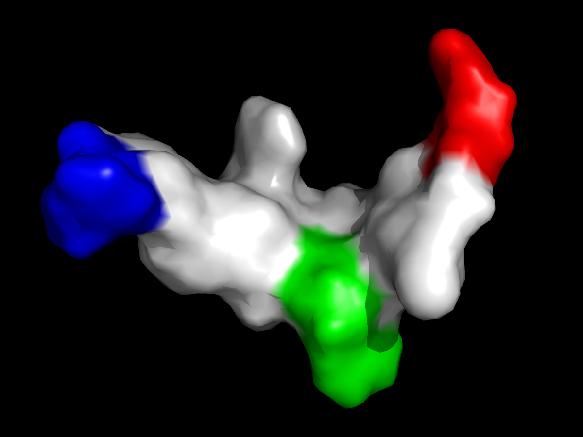
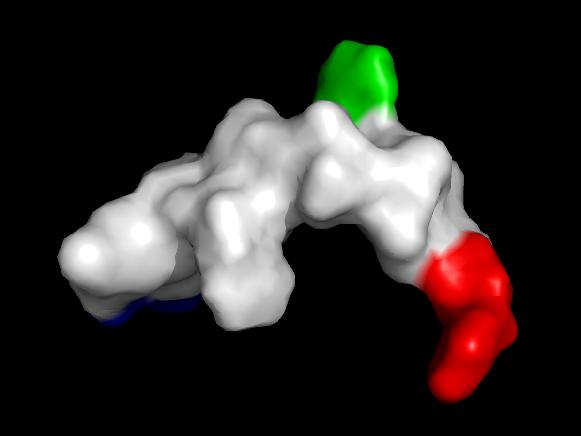
Färben oder kennzeichnen Sie 2-3 Aminosäuren Ihrer Wahl, um die Moleküle
besser vergleichen zu können. Vergleichen Sie die Oberflächen aus 3-4 Perspektiven:
stellen Sie mögliche Unterschiede der beiden Strukturen möglichst deutlich dar.
Makakensequenz RTD-1 (1HVZ) GFCRCLCRRGVCRCICTR
aus Pseudogen abgeleitete menschliche Sequenz Retrozyklin:! GICRCICGRGICRCICGR
Sequenz retro-Retrozyklin:! ! ! RGCICRCIGRGCICRCIG
Skript:
als pml: log.pml
Ergebnis:
Zunächst wurden die Aminosäuren von der Makakensequenz hin zur menschlichen Pseudoform mutiert. Anschließend wurden alle Aminosäuren invertiert.
Normalform / invertierte Pseudoform